System Golden Easy PCR (z barwnikiem)
Cechy
■ Dobra stabilność: Unikalna formuła sprawia, że cały układ reakcyjny jest bardzo stabilny. System zawiera składniki niezbędne do efektywnej amplifikacji PCR, takie jak termostabilna polimeraza DNA, dNTPs, MgCl2 i roztwór buforowy, a także różne specjalne stabilizatory, które znacznie zwiększają stabilność polimerazy i dNTP w normalnej temperaturze i 4 ℃.
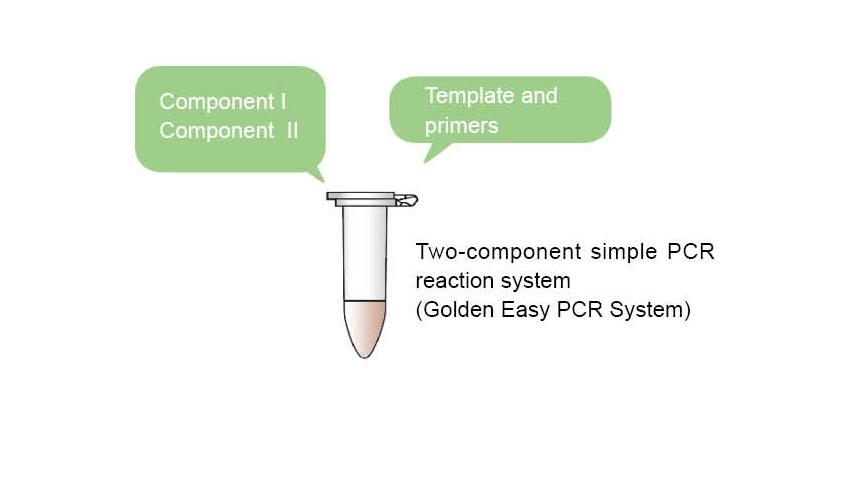
■ Szybki i prosty: Prosta i szybka obsługa. Po prostu zmieszaj oba składniki w proporcji, a następnie dodaj matryce i startery, aby przygotować reakcję, unikając żmudnych etapów dodawania różnych składników reakcji PCR jeden po drugim. Zminimalizuj błędy pobierania próbek i zanieczyszczenia krzyżowe, z niewielkim błędem między różnymi partiami i można je stosować w eksperymentach półilościowych.
■ Szerokie zastosowanie i wysoka czułość.
■ Każdy system reakcyjny zawiera barwniki, a elektroforezę można przeprowadzić bezpośrednio po etapach PCR, dzięki czemu procedura jest szybka i pracooszczędna.
Kontrola jakości
Czystość SDS-PAGE wynosi ponad 99%; Nie wykryto aktywności egzogennej nukleazy; Pojedynczy gen w ludzkim genomie może być skutecznie amplifikowany; Brak znaczącej zmiany aktywności przy przechowywaniu w temperaturze pokojowej przez tydzień.
Stabilność
Produkt przy użyciu dwuskładnikowego prostego systemu PCR można przechowywać w temperaturze 4°C przez jeden miesiąc bez widocznej zmiany aktywności.
Przepływ pracy
Krok 1: Wymieszaj proporcjonalnie różne składniki.
Krok 2: Natychmiast rozpocznij eksperyment.
Krok 3: Bezpośrednia elektroforeza dla mieszanki z barwnikami.
Krok 4: Zadowalające wyniki eksperymentalne.
Wszystkie produkty można dostosować do potrzeb ODM/OEM. Dla szczegółów,kliknij opcję Dostosowana usługa (ODM/OEM)
 |
Stosowany jest dwuskładnikowy prosty system PCR (Golden Easy PCR System). Układ reakcyjny to 50 μl (Jeżeli układy reakcji są różne należy proporcjonalnie zwiększyć lub zmniejszyć ilość składników reakcji odnoszących się do tego układu). |
 |
Stężenie końcowe reakcji |
Szablon A-1
■ Matryca zawiera zanieczyszczenia białkowe lub inhibitory Taq itp. ——Oczyść matrycę DNA, usuń zanieczyszczenia białkowe lub ekstrahuj matryce DNA za pomocą zestawów do oczyszczania.
■ Denaturacja szablonu nie jest kompletna —— Odpowiednio zwiększyć temperaturę denaturacji i przedłużyć czas denaturacji.
■ Degradacja szablonu — — Ponownie przygotuj szablon.
Podkład A-2
■ Słaba jakość starterów —— Ponownie zsyntetyzuj starter.
■ Degradacja primera —— Rozdzielić primery o wysokim stężeniu do małej objętości w celu konserwacji. Unikaj wielokrotnego zamrażania i rozmrażania lub długotrwałego kriokonserwacji w temperaturze 4°C.
■ Niewłaściwe zaprojektowanie starterów (np. niewystarczająca długość startera, dimer utworzony między starterami itp.) - Przeprojektowanie starterów (unikaj tworzenia dimeru startera i struktury drugorzędowej)
A-3 Mg2+stężenie
■ Mg2+ stężenie jest zbyt niskie ——Właściwie zwiększyć Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-4 Temperatura wyżarzania
■ Wysoka temperatura annealingu wpływa na wiązanie startera i matrycy. —— Zmniejsz temperaturę wyżarzania i zoptymalizuj warunki z gradientem 2°C.
A-5 Czas przedłużenia
■ Krótki czas przedłużenia——Wydłużenie czasu przedłużenia.
Zjawiska: Próbki ujemne również wykazują prążki sekwencji docelowej.
A-1 Zanieczyszczenie PCR
■ Zanieczyszczenie krzyżowe sekwencji docelowej lub produktów amplifikacji ——Uważaj, aby nie odpipetować próbki zawierającej sekwencję docelową w próbce ujemnej ani nie wylać jej z probówki wirówkowej. Odczynniki lub sprzęt powinny być autoklawowane w celu wyeliminowania istniejących kwasów nukleinowych, a zanieczyszczenie powinno być stwierdzone poprzez eksperymenty z kontrolą ujemną.
■ Zanieczyszczenie odczynnikiem —— Podziel odczynniki i przechowuj w niskiej temperaturze.
A-2 Primer
■ Mg2+ stężenie jest zbyt niskie ——Właściwie zwiększyć Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
■ Niewłaściwy projekt startera i sekwencja docelowa ma homologię z sekwencją inną niż docelowa. —— Przeprojektuj podkłady.
Zjawiska: Prążki amplifikacji PCR są niezgodne z oczekiwaną wielkością, albo duże, albo małe, lub czasami występują zarówno specyficzne prążki amplifikacji, jak i nieswoiste prążki amplifikacji.
Podkład A-1
■ Słaba specyficzność startera
—— Przeprojektuj podkład.
■ Stężenie podkładu jest zbyt wysokie ——Właściwie zwiększ temperaturę denaturacji i przedłuż czas denaturacji.
A-2 Mg2+ stężenie
■ Mg2+ stężenie jest zbyt wysokie ——Właściwie zmniejsz stężenie Mg2+: Zoptymalizuj Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-3 Termostabilna polimeraza
■ Nadmierna ilość enzymu —— Odpowiednio zmniejsz ilość enzymu w odstępach 0,5 U.
A-4 Temperatura wyżarzania
■ Temperatura wyżarzania jest zbyt niska —— Odpowiednio zwiększyć temperaturę wyżarzania lub zastosować metodę wyżarzania dwuetapowego
A-5 cykli PCR
■ Za dużo cykli PCR —— Zmniejsz liczbę cykli PCR.
Podkład A-1——Słaba specyficzność ——Przeprojektuj starter, zmień położenie i długość startera, aby wzmocnić jego specyficzność; lub wykonaj zagnieżdżoną reakcję PCR.
A-2 Szablon DNA
—— Matryca nie jest czysta —— Oczyść matrycę lub wyodrębnij DNA za pomocą zestawów do oczyszczania.
A-3 Mg2+ stężenie
——Mg2+ stężenie jest zbyt wysokie ——Właściwie zmniejsz Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-4 dNTP
—— Stężenie dNTP jest zbyt wysokie —— Odpowiednio zmniejsz stężenie dNTP
A-5 Temperatura wyżarzania
——Zbyt niska temperatura wyżarzania ——Odpowiednio zwiększyć temperaturę wyżarzania
A-6 cykli
——Zbyt wiele cykli ——Zoptymalizuj liczbę cykli

Pierwszym krokiem jest wybór odpowiedniej polimerazy. Zwykła polimeraza Taq nie może dokonać korekty ze względu na brak aktywności 3'-5' egzonukleazy, a niedopasowanie znacznie zmniejszy wydajność wydłużania fragmentów. Dlatego zwykła polimeraza Taq nie może skutecznie amplifikować fragmentów docelowych większych niż 5 kb. Polimerazę Taq ze specjalną modyfikacją lub inną polimerazę o wysokiej wierności należy wybrać w celu poprawy wydajności wydłużania i zaspokojenia potrzeb amplifikacji długich fragmentów. Ponadto amplifikacja długich fragmentów wymaga również odpowiedniego dostosowania projektu startera, czasu denaturacji, czasu wydłużania, pH buforu itp. Zwykle startery o wielkości 18-24 pz mogą prowadzić do lepszej wydajności. Aby zapobiec uszkodzeniu matrycy, czas denaturacji w 94°C należy skrócić do 30 sekund lub mniej na cykl, a czas wzrostu temperatury do 94°C przed amplifikacją powinien być krótszy niż 1 min. Ponadto ustawienie temperatury wydłużania na około 68°C i zaprojektowanie czasu wydłużania na poziomie 1 kb/min może zapewnić skuteczną amplifikację długich fragmentów.
Wskaźnik błędu amplifikacji PCR można zmniejszyć, stosując różne polimerazy DNA o wysokiej wierności. Spośród wszystkich dotychczas znalezionych polimeraz DNA Taq, enzym Pfu ma najniższy wskaźnik błędów i najwyższą wierność (patrz załączona tabela). Oprócz selekcji enzymów, naukowcy mogą jeszcze bardziej zmniejszyć szybkość mutacji PCR poprzez optymalizację warunków reakcji, w tym optymalizację składu buforu, stężenia termostabilnej polimerazy i optymalizację liczby cykli PCR.
Kategorie produktów
DLACZEGO WŁAŚNIE MY
Od momentu powstania nasza fabryka opracowuje produkty pierwszej światowej klasy, przestrzegając zasady
najpierw jakość. Nasze produkty zyskały doskonałą renomę w branży i cenne zaufanie wśród nowych i starych klientów.








