Zestaw DNA do plam krwi TIANamp
Cechy
■ Szerokie zastosowanie: Zestaw może być używany do bezpośredniej ekstrakcji gDNA z różnych próbek krwi.
■ Wysoka jakość: Dzięki unikalnemu systemowi buforów do lizy oczyszczone DNA o wysokim stężeniu, czystości i dobrej integralności może spełnić wymagania hybrydyzacji chipów i wysokoprzepustowego sekwencjonowania.
■ Szybki i nietoksyczny: zestaw wykorzystuje technologię adsorpcji na membranie krzemionkowej i nie wymaga fenolu ani chloroformu. Cały proces ekstrakcji można było zakończyć w ciągu godziny.
Aplikacje
Nadaje się do różnych regularnych zastosowań, takich jak trawienie enzymami restrykcyjnymi, PCR, konstrukcja biblioteki, Southern blot, hybrydyzacja chipów i sekwencjonowanie wysokoprzepustowe itp.
Wszystkie produkty można dostosować do potrzeb ODM/OEM. Dla szczegółów,kliknij opcję Dostosowana usługa (ODM/OEM)
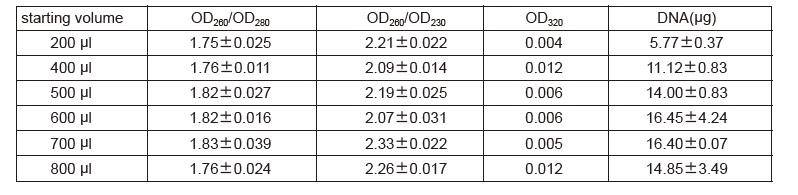
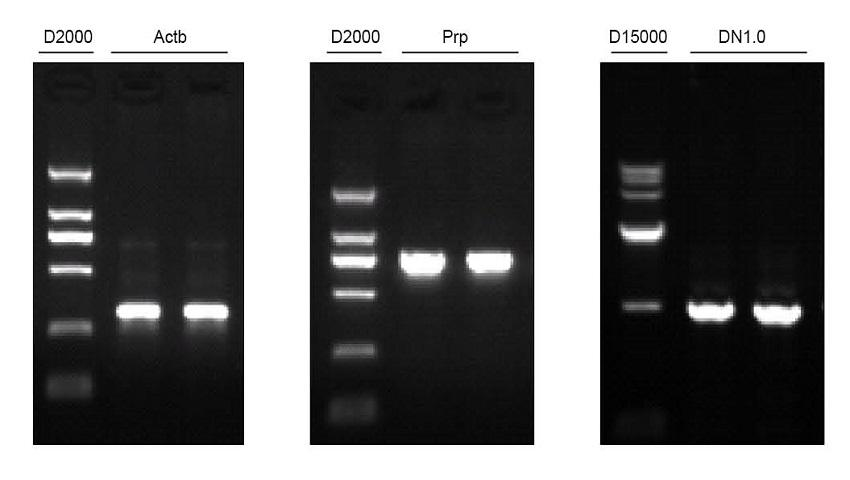
Genomowy DNA próbek ludzkiej krwi z kropli wyekstrahowano przy użyciu zestawu TIANamp Blood Spots DNA Kit zgodnie ze standardowym protokołem. Wyjściowa ilość próbki: 3 sztuki skrzepów krwi 3 x 3 mm. Jako matrycę PCR zastosowano 2 μl z 50 μl eluentów. Wykonać amplifikację PCR Actb (300 pz), PrP (750 pz) i DN1.0 (1 kb) odpowiednio w 20 μl systemie reakcyjnym PCR. Na linię wprowadzono 5 μl produktów PCR.
A-1 Niskie stężenie komórek lub wirusa w wyjściowej próbce — Wzbogać stężenie komórek lub wirusów.
A-2 Niewystarczająca liza próbek — Próbki nie zostały dokładnie wymieszane z buforem do lizy. Sugeruje się dokładne wymieszanie przez 1-2 krotne worteksowanie pulsacyjne. —Niewystarczająca liza komórek spowodowana spadkiem aktywności proteinazy K. —Niewystarczająca liza komórek lub degradacja białka z powodu niewystarczającego czasu ciepłej kąpieli. Zaleca się pocięcie tkanki na małe kawałki i wydłużenie czasu kąpieli w celu usunięcia wszystkich pozostałości w lizacie.
A-3 Niewystarczająca adsorpcja DNA. — Przed przeniesieniem lizatu do kolumny wirowej nie dodano etanolu ani nie dodano etanolu o niskiej zawartości procentowej zamiast 100% etanolu.
A-4 Wartość pH buforu elucyjnego jest zbyt niska. — Dostosuj pH do wartości między 8,0-8,3.
Pozostały etanol w eluencie.
— W eluencie znajdują się resztki buforu płuczącego PW. Etanol można usunąć przez odwirowanie kolumny wirowej przez 3-5 min, a następnie umieszczenie w inkubatorze w temperaturze pokojowej lub 50℃ na 1-2 min.
A-1 Próbka nie jest świeża. —Wyekstrahować dodatnią próbkę DNA jako kontrolę w celu ustalenia, czy DNA w próbce uległo degradacji.
A-2 Niewłaściwa obróbka wstępna. — Spowodowane nadmiernym rozdrobnieniem ciekłego azotu, odzyskaniem wilgoci lub zbyt dużą ilością próbki.
Obróbka wstępna powinna być różna dla różnych próbek. W przypadku próbek roślin należy dokładnie zmielić w ciekłym azocie. W przypadku próbek zwierzęcych zhomogenizować lub dokładnie zmielić w ciekłym azocie. W przypadku próbek o ścianach komórkowych, które są trudne do rozbicia, takich jak bakterie G+ i drożdże, do rozbicia ścian komórkowych zaleca się użycie lizozymu, lyticase lub metod mechanicznych.
4992201/4992202 Zestaw Plant Genomic DNA Kit wykorzystuje metodę opartą na kolumnie, która wymaga chloroformu do ekstrakcji. Szczególnie nadaje się do różnych próbek roślin, a także suchego proszku roślinnego. Zestaw Hi-DNAsecure Plant Kit jest również oparty na kolumnie, ale nie wymaga ekstrakcji fenolem/chloroformem, dzięki czemu jest bezpieczny i nietoksyczny. Nadaje się do roślin o wysokiej zawartości polisacharydów i polifenoli. 4992709/4992710 DNAquick Plant System wykorzystuje metodę na bazie cieczy. Nie jest również potrzebna ekstrakcja fenol/chloroform. Procedura oczyszczania jest prosta i szybka, bez ograniczeń dla ilości początkowych próbki, dzięki czemu użytkownicy mogą elastycznie dostosowywać ilość zgodnie z wymaganiami eksperymentalnymi. Duże rozmiary fragmentów gDNA można uzyskać z wysoką wydajnością.
Ekstrakcję DNA skrzepu krwi można przeprowadzić przy użyciu odczynników dostarczonych w tych dwóch zestawach, po prostu zmieniając protokół na specyficzną instrukcję ekstrakcji DNA skrzepu krwi. Miękka kopia protokołu ekstrakcji DNA skrzepu krwi może zostać wydana na żądanie.
Zawiesić świeżą próbkę w 1 ml PBS, soli fizjologicznej lub buforu TE. Całkowicie zhomogenizować próbkę za pomocą homogenizatora i zebrać osad na dnie probówki przez odwirowanie. Usunąć supernatant i ponownie zawiesić osad w 200 µl buforu GA. Poniższe oczyszczanie DNA można przeprowadzić zgodnie z instrukcją.
Do oczyszczania gDNA w próbkach osocza, surowicy i płynów ustrojowych zalecany jest zestaw TIANamp Micro DNA. Do oczyszczania gDNA wirusa z próbek surowicy/osocza zalecany jest zestaw TIANamp Virus DNA/RNA Kit. Do oczyszczania bakteryjnego gDNA z próbek surowicy i osocza zalecany jest zestaw TIANamp Bacteria DNA Kit (w przypadku dodatnich bakterii należy dołączyć lizozym). W przypadku próbek śliny zalecane są zestawy Hi-Swab DNA Kit i TIANamp Bacteria DNA Kit.
Do ekstrakcji genomu grzybów zalecane są zestawy DNAsecure Plant Kit lub DNAquick Plant System. Do ekstrakcji genomu drożdży zalecany jest zestaw TIANamp Yeast DNA Kit (litykaza powinna być przygotowana samodzielnie).
Kategorie produktów
DLACZEGO WŁAŚNIE MY
Od momentu powstania nasza fabryka opracowuje produkty pierwszej światowej klasy, przestrzegając zasady
najpierw jakość. Nasze produkty zyskały doskonałą renomę w branży i cenne zaufanie wśród nowych i starych klientów.