Zestaw do analizy HRM (EvaGreen)
Cechy
■ Wysoka rozdzielczość: Zastosuj nasycone barwniki EvaGreen, które mają wysoką rozdzielczość krzywej topnienia w stanie nasyconym i potrafią rozróżnić zmienność pojedynczej zasady.
■ Wysoka specyficzność: Użyj zmodyfikowanej przez przeciwciała polimerazy DNA hotstart, aby zmniejszyć nieswoistą amplifikację i poprawić specyficzność.
■ Wysoka stabilność: Starannie zoptymalizowany system buforowy zwiększa stabilność krzywej topnienia i poprawia wiarygodność wyników.
■ Korekcja ROX: barwnik ROX jest pakowany oddzielnie, co jest bardziej elastyczne w użyciu i zapewnia dokładniejsze wyniki.
Specyfikacja
Typ: polimeraza Taq DNA modyfikowana przeciwciałami, EvaGreen.
Zastosowania: Znane typowanie SNP; Nieznane badanie przesiewowe SNP; Skanowanie nieznanych zmutowanych genów; Analiza PCR metylacji.
Wszystkie produkty można dostosować do potrzeb ODM/OEM. Dla szczegółów,kliknij opcję Dostosowana usługa (ODM/OEM)
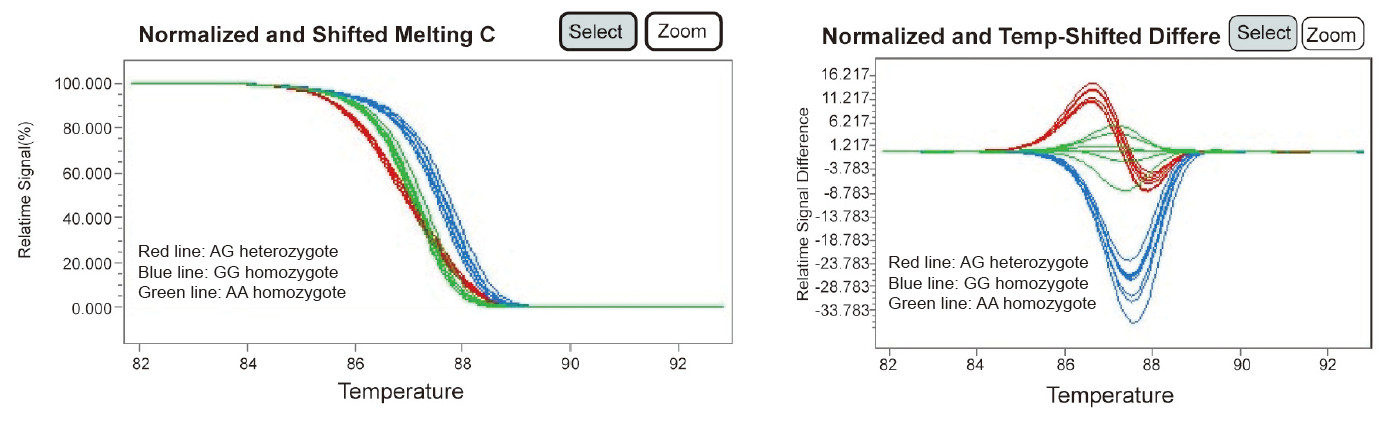
Genomowy DNA wyekstrahowano ze 100 μl próbki krwi ludzkiej przy użyciu zestawu TIANamp Blood DNA Kit. Do detekcji PCR w czasie rzeczywistym załadowano 50 ng DNA. Zgodnie z biblioteką NCBI SNP, znany SNP genu FEN1 (RS 174538) jest wybierany i wykrywany przy użyciu Roche LightCycle480. Wyniki są następujące:
Wyniki pokazują, że HRM Analysis Kit jest idealną metodą analizy locus SNP z wysoką powtarzalnością krzywej topnienia tego samego genotypu, wyraźną rozdzielczością różnych genotypów i bez pomyłek.
Informacje o SNP: ……CGGAAGAACACGTCG[A/G]CAGGAGCAGGCGCCT…… (Częstotliwość alleli: A=0,314, G=0,686)
Starter: Dla fragmentu 108 pz (FEN1_F: CCCTCAACGCTCTCACCATTTTG; FEN1_R: GGCACTTCCTTTCCGGTTGTG)
Kategorie produktów
DLACZEGO WŁAŚNIE MY
Od momentu powstania nasza fabryka opracowuje produkty pierwszej światowej klasy, przestrzegając zasady
najpierw jakość. Nasze produkty zyskały doskonałą renomę w branży i cenne zaufanie wśród nowych i starych klientów.