FastKing gDNA rozpraszający RT SuperMix
Cechy
■ Szybko: Jednoetapowe zakończenie usuwania genomu i wydajna odwrotna transkrypcja w ciągu 18 minut poprzez dodanie tylko szablonów.
■ Wysoka wydajność: Odwrotna transkryptaza jest modyfikowana motywem hydrofobowym, z wydajnością RT przekraczającą 95%.
■ Prosty i łatwy: wyjątkowa termoczuła DNaza ma szybki efekt, wysoką wydajność przy krótszym czasie reakcji i nie wpływa na cDNA.
Specyfikacja
Rodzaj: Odwrotna transkryptaza modyfikowana genem, gDNaza
Procedury: Jednoetapowy (usunięcie genomowego DNA i RT)
Wydajność RT: >95%
Szablon: 1 ng - 2 μg
Czas operacji: ~18 min
Aplikacje: Odwrotnie transkrybowany cDNA może być stosowany w konwencjonalnej reakcji PCR, PCR w czasie rzeczywistym, konstrukcji biblioteki cDNA, SAGE (Serial Analysis of Gene Expression), wydłużaniu startera i innych konwencjonalnych eksperymentach.
Wszystkie produkty można dostosować do potrzeb ODM/OEM. Dla szczegółów,kliknij opcję Dostosowana usługa (ODM/OEM)
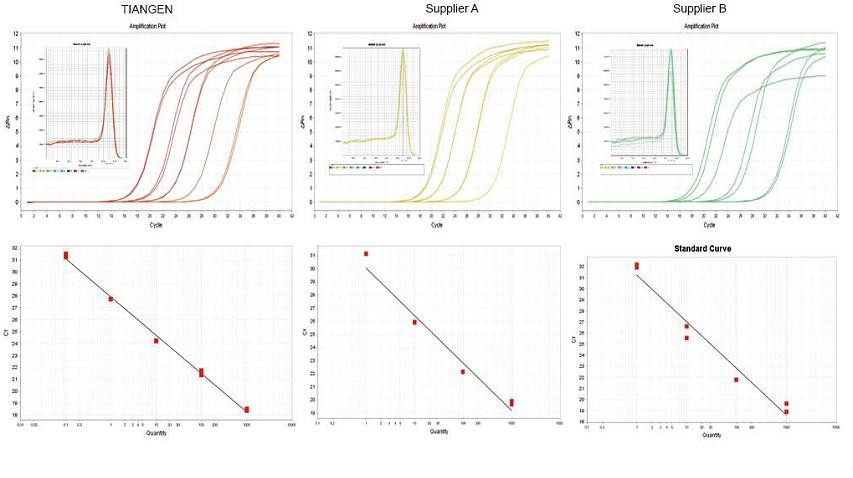 |
Przykład doświadczalny 1. cDNA zsyntetyzowano przy użyciu jednoetapowych odczynników do odwrotnej ilościowości TIANGEN FastKing gDNA Dispelling RT SuperMix, odpowiednich produktów odpowiednio od Dostawcy A i Dostawcy B. Wykrywano gen RN5 myszy stosując TIANGEN Talent qPCR PreMix (SYBR Green) i analizowano krzywą amplifikacji, krzywą topnienia i krzywą standardową. Wyniki pokazują, że TIANGEN FastKing gDNA Dispelling RT SuperMix ma najwyższą ilościową wartość Ct po odwrotnej transkrypcji i doskonałą odporność na stres oraz ma oczywiste zalety w przypadku matryc z wysokimi resztami zanieczyszczeń. |
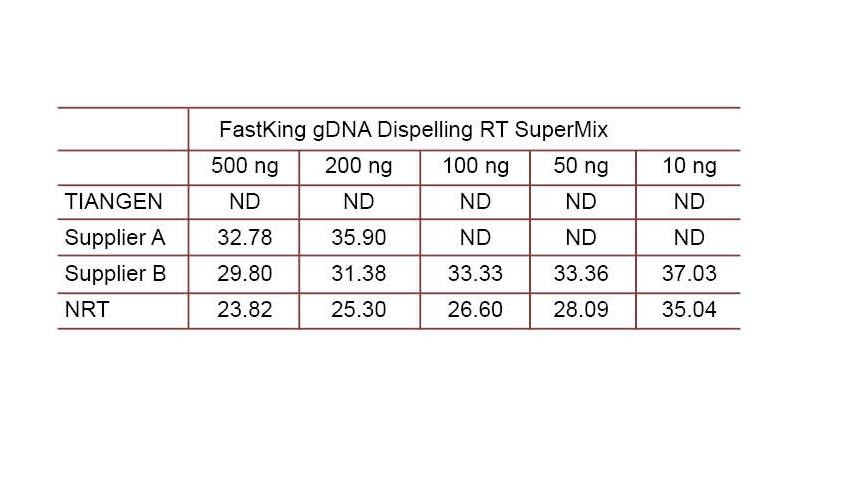 |
Przykład doświadczalny 2. cDNA zsyntetyzowano przy użyciu jednoetapowych odczynników do odwrotnej ilościowości z TIANGEN FastKing gDNA Dispelling RT SuperMix, odpowiednich produktów, odpowiednio, od Dostawcy A i Dostawcy B. Wykryj ludzki gen HsG za pomocą TIANGEN Talent qPCR PreMix (SYBR Green) i ręcznie dodaj różne stężenia genomowego DNA, aby wykryć zdolność usuwania gDNA różnych odczynników. Wyniki Ct pokazują, że TIANGEN FastKing gDNA Dispelling RT SuperMix ma doskonałą zdolność do usuwania genomowego DNA. Do 500 ng pozostałości genomowego DNA można doskonale usunąć bez wpływu na wyniki. ND: Nie wykryto. NRT: Wykrywanie mieszaniny bez odwrotnej transkrypcji. |
RNA A-1 ulega degradacji
——Oczyść wysokiej jakości RNA bez zanieczyszczeń. Materiał, z którego ekstrahuje się RNA, powinien być jak najświeższy, aby zapobiec degradacji RNA. Przeanalizuj integralność RNA na zdenaturowanym żelu przed reakcją RT. Po ekstrakcji RNA powinien być przechowywany w 100% formamidzie. Jeśli stosuje się inhibitor RNaz, temperatura ogrzewania powinna wynosić <45°C, a pH powinno być mniejsze niż 8,0, w przeciwnym razie inhibitor uwolni całą związaną RNazę. Ponadto inhibitor RNazy należy dodawać w roztworach zawierających ≥ 0,8 mM DTT.
A-2 RNA zawiera inhibitory reakcji odwrotnej transkrypcji
—— Inhibitory odwrotnej transkrypcji obejmują SDS, EDTA, glicerol, pirofosforan sodu, spermidynę, formamid, sól guanidyny itp. Zmieszaj kontrolny RNA z próbką i porównaj wydajność z reakcją kontrolnego RNA, aby sprawdzić, czy występuje inhibitor. Przemyć precypitację RNA 70% (v/v) etanolem w celu usunięcia inhibitorów.
A-3 Niewystarczające przyłączanie starterów użytych do syntezy pierwszej nici cDNA
——Ustal, czy temperatura wyżarzania jest odpowiednia dla starterów użytych w eksperymencie. W przypadku losowych heksamerów zaleca się utrzymywanie temperatury 25°C przez 10 min przed osiągnięciem temperatury reakcji. W przypadku starterów specyficznych dla genu (GSP), wypróbuj inny GSP lub przełącz się na oligo(dT) lub losowy heksamer.
A-4 Mała ilość początkowego RNA
——Zwiększ ilość RNA. W przypadku próbek RNA mniejszych niż 50 ng, w syntezie pierwszej nici cDNA można użyć 0,1 μg do 0,5 μg acetylo-BSA
A-5 Sekwencja docelowa nie ulega ekspresji w analizowanych tkankach.
——Wypróbuj inne chusteczki.
Reakcja PCR A-6 kończy się niepowodzeniem
——W przypadku dwuetapowej reakcji RT-PCR, matryca cDNA w etapie PCR nie może przekraczać 1/5 objętości reakcji.
A-1 Nieswoiste przyłączanie starterów i matryc
——Koniec 3' starterów nie powinien zawierać 2-3 dG lub dC. Użyj starterów specyficznych dla genu w syntezie pierwszej nici zamiast losowych starterów lub oligo(dT). W pierwszych kilku cyklach należy zastosować wyższą temperaturę wyżarzania, a następnie niższą temperaturę wyżarzania. Użyj polimerazy DNA Taq typu hot start do PCR, aby poprawić specyficzność reakcji.
A-2 Słaba konstrukcja starterów specyficznych dla genu
—— Postępuj zgodnie z tymi samymi zasadami projektowania starterów amplifikacji.
RNA A-3 zanieczyszczone genomowym DNA
—— Traktuj RNA DNazą I klasy PCR. Skonfiguruj reakcję kontrolną bez odwrotnej transkrypcji, aby wykryć zanieczyszczenie DNA.
A-4 Formowanie dimeru podkładowego
——Zaprojektuj startery bez sekwencji komplementarnych na końcu 3'.
A-5 Zbyt wysoki Mg2+ stężenie
—— Optymalizacja Mg2+ stężenie dla każdej kombinacji matrycy i startera
A-6 Zanieczyszczone obcym DNA
——Używaj końcówek odpornych na aerozol i enzymów UDG.
A-1 Zawartość produktu pierwszej nitki jest zbyt wysoka
—— Zmniejsz ilość produktu pierwszej nici w konwencjonalnym etapie reakcji PCR.
A-2 Zbyt duża ilość startera w reakcji PCR
—— Zmniejsz ilość wprowadzanych starterów.
A-3 Za dużo cykli
——Zoptymalizuj warunki reakcji PCR i zmniejsz liczbę cykli PCR.
A-4 Zbyt niska temperatura wyżarzania
—— Zwiększyć temperaturę wyżarzania, aby zapobiec niespecyficznej inicjacji i wydłużeniu.
A-5 Nieswoista amplifikacja fragmentów oligonukleotydów wytworzonych przez degradację DNA przez DNazę — — Ekstrakcja wysokiej jakości RNA, aby zapobiec zanieczyszczeniu DNA.
RT-PCR ma na celu odwrotną transkrypcję RNA do cDNA, a następnie użycie cDNA poddanego odwrotnej transkrypcji jako matrycy do reakcji PCR w celu amplifikacji docelowego fragmentu. Wybierz losowe startery, Oligo dT lub startery specyficzne dla genu zgodnie z określonymi warunkami eksperymentu. Wszystkie powyższe startery można stosować do mRNA krótkich komórek eukariotycznych bez struktury spinki do włosów.
Starter losowy: odpowiedni dla długich RNA o strukturze spinki do włosów, a także wszystkich rodzajów RNA, takich jak rRNA, mRNA, tRNA itp. Są one głównie używane do reakcji RT-PCR z pojedynczą matrycą.
Oligo dT: Nadaje się do RNA z ogonami PolyA (prokariotyczne RNA, eukariotyczne Oligo dT rRNA i tRNA nie mają ogonów PolyA). Ponieważ Oligo dT jest związany z ogonem PolyA, jakość próbek RNA musi być wysoka, a nawet niewielka ilość degradacji znacznie zmniejszy ilość pełnej długości syntezy cDNA.
Starter specyficzny dla genu: Komplementarny do sekwencji matrycy, odpowiedni w sytuacjach, w których znana jest sekwencja docelowa.
Istnieją dwa sposoby:
1. Wewnętrzna metoda referencyjna: teoretycznie cDNA to fragmenty DNA o różnej długości, więc wynikiem elektroforezy jest rozmaz. Jeśli liczebność RNA jest niska, żaden produkt nie pojawi się w elektroforezie, ale nie oznacza to, że żaden produkt nie zostanie zamplifikowany przez PCR. Ogólnie do wykrywania cDNA można użyć wewnętrznego odniesienia. Jeśli wewnętrzne odniesienie ma wyniki, jakość cDNA można w zasadzie zagwarantować (w kilku przypadkach, jeśli docelowy fragment genu jest zbyt długi, mogą wystąpić wyjątki).
2. Jeśli istnieje znany gen amplifikowany przez tę matrycę, można go zweryfikować za pomocą starterów tego genu. Amplifikacja wewnętrznego odniesienia niekoniecznie oznacza, że nie ma problemu z cDNA. Ponieważ wewnętrzne odniesienie ma dużą ilość cDNA, łatwo je amplifikować. Jeśli cDNA zostanie częściowo zdegradowany z różnych powodów, z punktu widzenia prawdopodobieństwa, wyniki PCR dla genów docelowych o niskiej liczebności będą miały duży wpływ. Podczas gdy wewnętrzne odniesienie jest nadal obfite, wzmocnienie prawdopodobnie nie zostanie naruszone.
Częściowa degradacja RNA. Wykryj integralność i oczyść RNA
Zawartość RNA różnych gatunków może być różna, ale ogólnie wyekstrahowany całkowity RNA powinien zawierać dwa czyste prążki 28S i 18S w elektroforezie żelowej, a jasność pierwszego prążka powinna być dwa razy większa niż drugiego. Pasmo 5S wskazuje, że RNA uległ degradacji, a jego jasność jest proporcjonalna do stopnia degradacji. Pomyślna amplifikacja wzorca wewnętrznego nie oznacza, że nie ma problemu z RNA, ponieważ wzorzec wewnętrzny występuje w dużej ilości, RNA może być amplifikowane, o ile degradacja nie jest poważna. OD260/OD280stosunek czystego RNA mierzony spektrofotometrem powinien wynosić od 1,9 do 2,1. Niewielka ilość zanieczyszczeń białkowych w RNA zmniejszy ten stosunek. Dopóki wartość nie jest zbyt niska, nie będzie to miało wpływu na RT. Najważniejsza dla RT jest integralność RNA.
Wydłużenie wewnętrznego genu odniesienia może jedynie wskazywać, że RT się powiodło, ale niekoniecznie jest związane z jakością nici cDNA. Ponieważ wewnętrzne fragmenty odniesienia mają na ogół mały rozmiar i wysoką ekspresję, łatwiej jest im odnieść sukces w odwrotnej transkrypcji. Jednak wielkość i ekspresja genu docelowego różni się w zależności od genu. Jakości cDNA nie można ocenić tylko na podstawie wewnętrznego odniesienia, zwłaszcza dla fragmentów docelowych dłuższych niż 2 kb.
Niektóre próbki mają złożone struktury drugorzędowe, mają bogatą zawartość GC lub są cenne w niskiej liczebności. W takich przypadkach należy wybrać odpowiednią odwrotną transkryptazę w zależności od wielkości docelowego fragmentu i próbki. W przypadku matryc RNA o wysokiej zawartości GC i złożonej strukturze drugorzędowej, trudno jest otworzyć strukturę drugorzędową w niskiej temperaturze lub za pomocą zwykłej odwrotnej transkryptazy. W przypadku tych matryc można wybrać odwrotną transkryptazę Quant, ponieważ jej wydajność odwrotnej transkrypcji jest oczywiście lepsza niż odwrotnej transkryptazy serii M-MLV, która może wydajnie odwrotną transkrypcję różnych matryc RNA i w maksymalnym stopniu dokonać transkrypcji RNA do pierwszej nici cDNA. Przy użyciu ogólnego zestawu do odwrotnej transkryptazy system 20 μl może skutecznie odwrócić transkrypcję tylko 1 μg całkowitego RNA. Proszę zwrócić uwagę na maksymalną pojemność RT zestawu. Jeśli matryca zostanie dodana w nadmiarze, odwrotna transkrypcja będzie faworyzować RNA z dużą ilością. Dlatego lepiej nie przekraczać maksymalnej wydajności systemu.
A-1 Określ, czy RNA jest poważnie zdegradowany i czy RT jest skuteczne
Ogólnie rzecz biorąc, przyczyną niepowodzenia wewnętrznej amplifikacji referencyjnej jest często poważna degradacja RNA. Innym możliwym powodem jest niepowodzenie odwrotnej transkrypcji. Odniesienia wewnętrznego nie można użyć jako standardu do oceny jakości pojedynczej nici cDNA, ale można go użyć jako standardu do oceny, czy odwrotna transkrypcja jest skuteczna, jeśli nie ma problemu z jakością RNA. Najważniejszą rzeczą w procesie odwrotnej transkrypcji jest utrzymanie stałej temperatury i stałego układu reakcyjnego w celu poprawy wydajności reakcji.
A-2 Określ, czy startery do amplifikacji wewnętrznych genów odniesienia są wiarygodne i czy są jakieś problemy z odczynnikami użytymi w reakcji PCR.
Dla względnej oceny ilościowej, RNA musi być oznaczane ilościowo przed odwrotną transkrypcją, co jest również wymagane w wielu zestawach do odwrotnej transkrypcji, na przykład, ilościowo RNA wejściowy jest określany jako 1 μg. Ponieważ cDNA poddany odwrotnej transkrypcji jest roztworem mieszanym, zawierającym RNA, oligo dT, enzym, dNTP, a nawet niewielką ilość reszty DNA, spowoduje to odchylenie, więc nie jest możliwe dokładne określenie ilościowe cDNA. Dlatego konieczna jest kwantyfikacja RNA. Biorąc pod uwagę, że wydajność odwrotnej transkrypcji jest taka sama w różnych próbkach, ilość otrzymanego cDNA powinna być taka sama, a analiza ilościowa może pokazać porównanie poziomów ekspresji różnych genów w tej samej ilości całkowitego RNA. W przypadku przeprowadzania względnej fluorescencyjnej ilościowej PCR, ilościowe cDNA może nie być wymagane po odwrotnej transkrypcji, ponieważ wewnętrzny gen odniesienia może działać jako odniesienie.
Jest to głównie związane z genami, a odwrotna transkrypcja długiego fragmentu nie jest możliwa dla większości genów. Po pierwsze, wydajność odwrotnej transkrypcji jest znacznie niższa niż PCR. Po drugie, region bogaty w GC i drugorzędowa struktura wielu genów ogranicza zarówno odwrotną transkrypcję, jak i PCR. Wreszcie, wierność i wydajność amplifikacji PCR są trudne do jednoczesnego zagwarantowania. W procesie odwrotnej transkrypcji nikt nie może zagwarantować uzyskania długiego fragmentu dla genów o niskiej liczbie kopii, zwłaszcza przy użyciu oligo dT. Jeśli chodzi o 5' UTR z większą ilością GC, jest jeszcze trudniej. Dlatego nadal rozsądną metodą jest odwrotna transkrypcja za pomocą losowych starterów, znalezienie naturalnych miejsc cięcia we fragmencie docelowym, amplifikacja przez segmenty, a następnie przeprowadzenie trawienia restrykcyjnego i ligacji. Generalnie trudno jest bezpośrednio amplifikować fragmenty większe niż 2 kb, ale nie zawsze jest możliwe uzyskanie: 1. Przede wszystkim zagwarantowanie integralności RNA/mRNA, preferowana jest ekstrakcja TRIZOL. 2. Zestaw M-MLV RT-PCR może być używany bezpośrednio. Wydłuż czas wyżarzania i odpowiednio zwiększ liczbę cykli w procesie amplifikacji. Alternatywnie można zastosować zagnieżdżoną reakcję PCR lub przeprowadzić najpierw jedną lub dwie reakcje z odpowiednio wydłużonym czasem denaturacji i wydłużania przed normalną amplifikacją PCR, co może pomóc w wydłużaniu fragmentów. Zwróć uwagę na wierność polimerazy. 3.Long Taq może być użyty w PCR, aby uzyskać idealne wyniki. 4. Do aplikacji ekspresji białek należy zastosować polimerazę o wysokiej wierności.
Istnieją dwa rodzaje odwrotnej transkryptazy oferowane przez TIANGEN: Quant/King RTase i TIANScript M-MLV. Główną różnicą między nimi jest ilość wprowadzanych szablonów. Quant jest unikalną odwrotną transkryptazą, która różni się od powszechnie stosowanej M-MLV pochodzącej z wirusa mysiej białaczki Moloneya. Quant to nowa, wysoce wydajna odwrotna transkryptaza ulegająca rekombinacji, wyrażana przez inżynierię Escherichia coli. Quant jest odpowiedni do amplifikacji 50 ng-2 μg RNA o wysokiej aktywności odwrotnej transkrypcji i wysokiej wydajności. W porównaniu ze zwykłymi MMLV lub AMV, największą cechą Quant jest to, że ma bardzo silne powinowactwo do matryc RNA i może odwrócić złożone matryce transkrypcji bez denaturacji w wysokiej temperaturze. W przypadku szablonów o wyższej zawartości GC, wydajność odwrotna jest wyższa. Jednak ta odwrotna transkryptaza ma aktywność RNazy H, która może wpływać na długość produktu cDNA (odpowiednia dla matryc < 4,5 kb). Do konwencjonalnej odwrotnej transkrypcji zaleca się odwrotną transkryptazę TIANScript MMLV. Ta RTaza jest zmodyfikowanym enzymem o bardzo słabej aktywności RNazy H, który jest odpowiedni do długiej (> 5 kb) syntezy cDNA.
Jednoetapowa odwrotna transkrypcja i amplifikacja PCR są wykonywane w tej samej probówce bez otwierania pokrywy probówki pomiędzy syntezą cDNA a amplifikacją, co jest pomocne w zmniejszeniu zanieczyszczenia. Ponieważ wszystkie otrzymane próbki cDNA są używane do amplifikacji, czułość jest wyższa i wynosi minimum 0,01 pg całkowitego RNA. W przypadku udanego jednoetapowego RTPCR do inicjowania syntezy cDNA na ogół stosuje się startery specyficzne dla genu. Metoda dwuetapowa, a mianowicie odwrotna transkrypcja i amplifikacja PCR odbywa się w dwóch etapach. Najpierw przeprowadza się odwrotną transkrypcję z matrycy RNA w celu uzyskania cDNA, a otrzymany cDNA poddaje się jednej lub większej liczbie różnych reakcji PCR. Metoda dwuetapowa może wykorzystywać startery oligo(dT) lub losowe do prowadzenia syntezy pierwszej nici cDNA i może odwrócić transkrypcję wszystkich informacji o mRNA z określonej próbki.
Kategorie produktów
DLACZEGO WŁAŚNIE MY
Od momentu powstania nasza fabryka opracowuje produkty pierwszej światowej klasy, przestrzegając zasady
najpierw jakość. Nasze produkty zyskały doskonałą renomę w branży i cenne zaufanie wśród nowych i starych klientów.