Zestaw do bezpośredniego PCR z krwi
Cechy
■ Prosty i szybki: amplifikację PCR można przeprowadzić bezpośrednio z użyciem krwi jako matrycy, bez konieczności żmudnych etapów przygotowania próbki i ekstrakcji DNA.
■ Wysoka czystość: Pominięcie etapów wstępnego przygotowania próbki i ekstrakcji DNA może pomóc w uniknięciu krzyżowego zanieczyszczenia próbek.
■ Wysoka przepustowość: Identyfikację PCR próbek na dużą skalę można przeprowadzić przez połączenie zestawu z 96/384-dołkowymi płytkami do PCR.
■ Silna uniwersalność: Ten zestaw może wydajnie amplifikować fragmenty o wysokiej GC lub fragmenty o złożonej strukturze drugorzędowej, a długość amplifikacji może wynosić do 5 kb.
■ Duża odporność na stres: Ten zestaw może być stosowany do różnych gatunków i próbek krwi zakonserwowanych na różne sposoby.
Aplikacje
Produkty PCR z tego zestawu zawierają „A” na końcu 3', co może być bezpośrednio użyte do klonowania wektora TA. Zestaw ten może być używany do amplifikacji fragmentów genomowego DNA, wysokoprzepustowej analizy genetycznej i analizy genotypowania (np. wykrywania genów).
Wszystkie produkty można dostosować do potrzeb ODM/OEM. Dla szczegółów,kliknij opcję Dostosowana usługa (ODM/OEM)
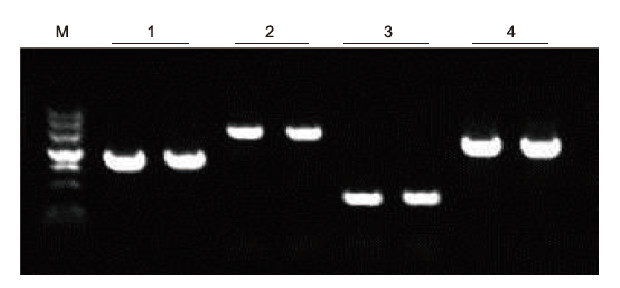 |
Stosując ludzką antykoagulację EDTA jako matrycę, 4 geny o różnej zawartości GC amplifikowano za pomocą zestawu Blood Direct PCR Kit. System reakcyjny PCR wynosił 20 μl, a jako matrycę stosowano 1 μl krwi. M: Znacznik TIANGEN II; 1: Wielkość fragmentu 1090 pz, zawartość GC 68,1%; 2: Wielkość fragmentu 1915 pz, zawartość GC 70,4%; 3: Wielkość fragmentu 448 bp, zawartość GC 74,8%; 4: Wielkość fragmentu 1527 bp, zawartość GC 61,5%. Wyniki eksperymentalne: Zestaw Blood Direct PCR może skutecznie amplifikować fragmenty DNA z zawartością GC w zakresie 61,5%-74,8%, co sugeruje, że jest zdolny do amplifikacji fragmentów o wysokiej zawartości GC. |
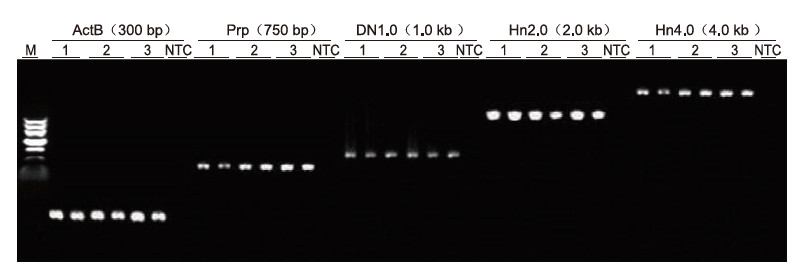 |
Stosując ludzką antykoagulację EDTA jako matrycę, 5 genów o różnych długościach (ActB, Prp, DN1.0, Hn2.0 i Hn4.0) zamplifikowano za pomocą zestawu Blood Direct PCR. System reakcyjny PCR wynosił 20 μl, a jako matrycę stosowano 1 μl krwi. M: Znacznik TIANGEN II; 1-3: 3 różne próbki krwi; NTC: kontrola bez starterów. Wyniki eksperymentalne: Zestaw Blood Direct PCR może amplifikować fragmenty o długości do 4 kb, co sugeruje, że jest zdolny do amplifikacji długich fragmentów. |
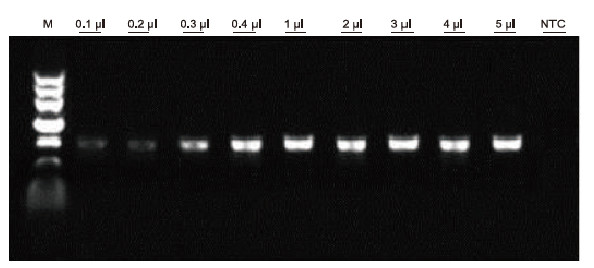 |
Stosując ludzką antykoagulację EDTA jako matrycę, do wykrywania metodą PCR różnych próbek krwi zastosowano zestaw Blood Direct PCR Kit. System reakcyjny PCR wynosił 20 μl, a jako matrycę stosowano 1 μl krwi. M: Znacznik TIANGEN II; 1-9: obciążona ilość krwi wynosi odpowiednio 0,1 μl, 0,2 μl, 0,3 μl, 0,4 μl, 1 μl, 2 μl, 3 μl, 4 μl i 5 μl; NTC: kontrola bez szablonu Wyniki eksperymentalne: Blood Direct PCR Kit ma dużą odporność na krew i może amplifikować próbki krwi w zakresie obciążenia 0,1-5 μl. |
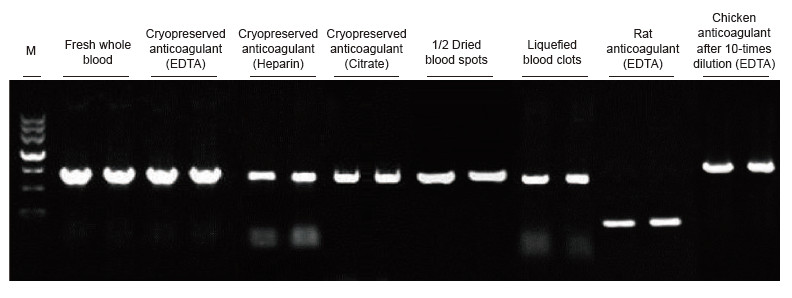 |
Jako matryce zastosowano próbki krwi człowieka, szczura, kurczaka i innych gatunków poddane różnym zabiegom. Do amplifikacji PRNP (ludzki, 750 pz), aktyny (szczur, 200 pz) i β-aktyny (kurczak, 1,0 kb) zastosowano zestaw Blood Direct PCR. System reakcyjny PCR wynosił 20 μl, a jako matrycę stosowano 1 μl krwi. M: Znacznik TIANGEN II. Wyniki eksperymentalne: Blood Direct PCR Kit może być stosowany do szerokiej gamy próbek, a bezpośrednia detekcja PCR może być przeprowadzana na próbkach krwi z różnych gatunków z różnymi zabiegami. |
Szablon A-1
■ Matryca zawiera zanieczyszczenia białkowe lub inhibitory Taq itp. ——Oczyść matrycę DNA, usuń zanieczyszczenia białkowe lub ekstrahuj matryce DNA za pomocą zestawów do oczyszczania.
■ Denaturacja szablonu nie jest kompletna —— Odpowiednio zwiększyć temperaturę denaturacji i przedłużyć czas denaturacji.
■ Degradacja szablonu — — Ponownie przygotuj szablon.
Podkład A-2
■ Słaba jakość starterów —— Ponownie zsyntetyzuj starter.
■ Degradacja primera —— Rozdzielić primery o wysokim stężeniu do małej objętości w celu konserwacji. Unikaj wielokrotnego zamrażania i rozmrażania lub długotrwałego kriokonserwacji w temperaturze 4°C.
■ Niewłaściwe zaprojektowanie starterów (np. niewystarczająca długość startera, dimer utworzony między starterami itp.) - Przeprojektowanie starterów (unikaj tworzenia dimeru startera i struktury drugorzędowej)
A-3 Mg2+stężenie
■ Mg2+ stężenie jest zbyt niskie ——Właściwie zwiększyć Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-4 Temperatura wyżarzania
■ Wysoka temperatura annealingu wpływa na wiązanie startera i matrycy. —— Zmniejsz temperaturę wyżarzania i zoptymalizuj warunki z gradientem 2°C.
A-5 Czas przedłużenia
■ Krótki czas przedłużenia——Wydłużenie czasu przedłużenia.
Zjawiska: Próbki ujemne również wykazują prążki sekwencji docelowej.
A-1 Zanieczyszczenie PCR
■ Zanieczyszczenie krzyżowe sekwencji docelowej lub produktów amplifikacji ——Uważaj, aby nie odpipetować próbki zawierającej sekwencję docelową w próbce ujemnej ani nie wylać jej z probówki wirówkowej. Odczynniki lub sprzęt powinny być autoklawowane w celu wyeliminowania istniejących kwasów nukleinowych, a zanieczyszczenie powinno być stwierdzone poprzez eksperymenty z kontrolą ujemną.
■ Zanieczyszczenie odczynnikiem —— Podziel odczynniki i przechowuj w niskiej temperaturze.
A-2 Primer
■ Mg2+ stężenie jest zbyt niskie ——Właściwie zwiększyć Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
■ Niewłaściwy projekt startera i sekwencja docelowa ma homologię z sekwencją inną niż docelowa. —— Przeprojektuj podkłady.
Zjawiska: Prążki amplifikacji PCR są niezgodne z oczekiwaną wielkością, albo duże, albo małe, lub czasami występują zarówno specyficzne prążki amplifikacji, jak i nieswoiste prążki amplifikacji.
Podkład A-1
■ Słaba specyficzność startera
—— Przeprojektuj podkład.
■ Stężenie podkładu jest zbyt wysokie ——Właściwie zwiększ temperaturę denaturacji i przedłuż czas denaturacji.
A-2 Mg2+ stężenie
■ Mg2+ stężenie jest zbyt wysokie ——Właściwie zmniejsz stężenie Mg2+: Zoptymalizuj Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-3 Termostabilna polimeraza
■ Nadmierna ilość enzymu —— Odpowiednio zmniejsz ilość enzymu w odstępach 0,5 U.
A-4 Temperatura wyżarzania
■ Temperatura wyżarzania jest zbyt niska —— Odpowiednio zwiększyć temperaturę wyżarzania lub zastosować metodę wyżarzania dwuetapowego
A-5 cykli PCR
■ Za dużo cykli PCR —— Zmniejsz liczbę cykli PCR.
Podkład A-1——Słaba specyficzność ——Przeprojektuj starter, zmień położenie i długość startera, aby wzmocnić jego specyficzność; lub wykonaj zagnieżdżoną reakcję PCR.
A-2 Szablon DNA
—— Matryca nie jest czysta —— Oczyść matrycę lub wyodrębnij DNA za pomocą zestawów do oczyszczania.
A-3 Mg2+ stężenie
——Mg2+ stężenie jest zbyt wysokie ——Właściwie zmniejsz Mg2+ stężenie: Optymalizacja Mg2+ stężenie poprzez serię reakcji od 1 mM do 3 mM w odstępie 0,5 mM w celu określenia optymalnego Mg2+ stężenie dla każdej matrycy i startera.
A-4 dNTP
—— Stężenie dNTP jest zbyt wysokie —— Odpowiednio zmniejsz stężenie dNTP
A-5 Temperatura wyżarzania
——Zbyt niska temperatura wyżarzania ——Odpowiednio zwiększyć temperaturę wyżarzania
A-6 cykli
——Zbyt wiele cykli ——Zoptymalizuj liczbę cykli

Pierwszym krokiem jest wybór odpowiedniej polimerazy. Zwykła polimeraza Taq nie może dokonać korekty ze względu na brak aktywności 3'-5' egzonukleazy, a niedopasowanie znacznie zmniejszy wydajność wydłużania fragmentów. Dlatego zwykła polimeraza Taq nie może skutecznie amplifikować fragmentów docelowych większych niż 5 kb. Polimerazę Taq ze specjalną modyfikacją lub inną polimerazę o wysokiej wierności należy wybrać w celu poprawy wydajności wydłużania i zaspokojenia potrzeb amplifikacji długich fragmentów. Ponadto amplifikacja długich fragmentów wymaga również odpowiedniego dostosowania projektu startera, czasu denaturacji, czasu wydłużania, pH buforu itp. Zwykle startery o wielkości 18-24 pz mogą prowadzić do lepszej wydajności. Aby zapobiec uszkodzeniu matrycy, czas denaturacji w 94°C należy skrócić do 30 sekund lub mniej na cykl, a czas wzrostu temperatury do 94°C przed amplifikacją powinien być krótszy niż 1 min. Ponadto ustawienie temperatury wydłużania na około 68°C i zaprojektowanie czasu wydłużania na poziomie 1 kb/min może zapewnić skuteczną amplifikację długich fragmentów.
Wskaźnik błędu amplifikacji PCR można zmniejszyć, stosując różne polimerazy DNA o wysokiej wierności. Spośród wszystkich dotychczas znalezionych polimeraz DNA Taq, enzym Pfu ma najniższy wskaźnik błędów i najwyższą wierność (patrz załączona tabela). Oprócz selekcji enzymów, naukowcy mogą jeszcze bardziej zmniejszyć szybkość mutacji PCR poprzez optymalizację warunków reakcji, w tym optymalizację składu buforu, stężenia termostabilnej polimerazy i optymalizację liczby cykli PCR.
Kategorie produktów
DLACZEGO WŁAŚNIE MY
Od momentu powstania nasza fabryka opracowuje produkty pierwszej światowej klasy, przestrzegając zasady
najpierw jakość. Nasze produkty zyskały doskonałą renomę w branży i cenne zaufanie wśród nowych i starych klientów.








